ゲノム編集技術を2つ組み合わせて長いDNAの挿入に成功! (スペインの最新研究)

健康情報
今回は長いDNA断片を位置特異的に挿入する新しい方法が発表されたので報告します。
スペイン バルセロナのIDIBELL医療研究センターが最新研究をプレスリリース(英文)しましたので、それを和訳したものをご紹介します。
ニュースタイトル「Nested CRISPRは長いDNAフラグメントを利用して効率的なゲノム編集を可能にする」
CRISPRは、高精度のゲノム編集を通じて生物医学研究に革命を起こしている技術です。しかしながら、たとえそれが単一または少数のヌクレオチドからなる突然変異の作成または修正を比較的容易にしても、それは依然としてゲノム中の大きなDNAフラグメントに対しては制限があります。例えば、広く使用されているGFPのような蛍光タンパク質を産生する遺伝子のゲノム挿入は効率が悪く、複雑なクローニング工程を伴います。Bellvitge Biomedical Research Institute(IDIBELL)のCerón博士のグループは、モデル生物の線虫を利用してこの手法を最適化し、Nested CRISPR(ネステッド・クリスパー)と呼ばれる手法の開発につながりました。このクローニングフリーの方法は、2つのステップで長いDNA断片を挿入することを含みます。最初のステップでは、長いフラグメントのごく一部(200ヌクレオチド未満)がゲノムに挿入されます。第二段階の間に、この小さな断片は(約1,000塩基の)より長い断片を効率的に挿入するための「ネスト」または「ランディングパッド」として機能します。
アメリカ遺伝学会のジャーナルである Genetics に最近発表されたこの論文は、これまでにない関心を呼んでいます。線虫のようにライフサイクルが短いモデル生物では、研究者はCRISPRの可能性と限界の両方を探ることができます。研究者であるJeremy Vicencio氏は、博士研究員のCarmenMartínez氏とXèniaSerrat氏と一緒に、Nested CRISPRの効率をしっかりと納得のいくように実証するために、線虫の生殖細胞系列での数百のマイクロインジェクションと、何千もの遺伝子型決定を行いました。
Nested CRISPRは、DNA修復の最初のステップに市販のオリゴヌクレオチドを、2番目のステップに一般的なPCR試薬を利用しています。これは、蛍光タンパク質で何百もの遺伝子をタグ付けすることを可能にすることによって大規模実験を容易にします。さらに、Nested CRISPRはモジュール式であるので、目的のさらなるペプチドまたはタンパク質もまたこれらの蛍光タンパク質と組み合わせて組み込むことができます。例えばこのグループは現在、これらの蛍光タンパク質を制御された方法でそれらの分解を可能にするペプチドでタグ付けすることに取り組んでいます。
最後に、IDIBELLのCerón博士によって進められた疾患モデル線虫は、Nested CRISPRを使用して線虫の遺伝子をヒトのものに置き換えることを意図しています。これは、疾患に関連するヒトの突然変異の影響を研究するための多細胞モデルとして、この小さな線虫の使用を促すでしょう。このシステムは、それが突然変異またはゲノム変異(多型として知られている)の病原性に関して迅速かつ効率的に予測することができるので、患者に合った医療おいて実質的な利益となるでしょう。
引用ニュース & 原著論文
🔵 英語ニュース:The novel method Nested CRISPR enables efficient genome editing using long DNA fragments(IDIBELL-Bellvitge Biomedical Research Institute )
🔵 元の学術論文:Efficient Generation of Endogenous Fluorescent Reporters by Nested CRISPR in Caenorhabditis elegans (GENETICS Early online January 31, 2019)
Seigoの追記
少し分かりずらかったので要点を発表されているに図に付け足しました。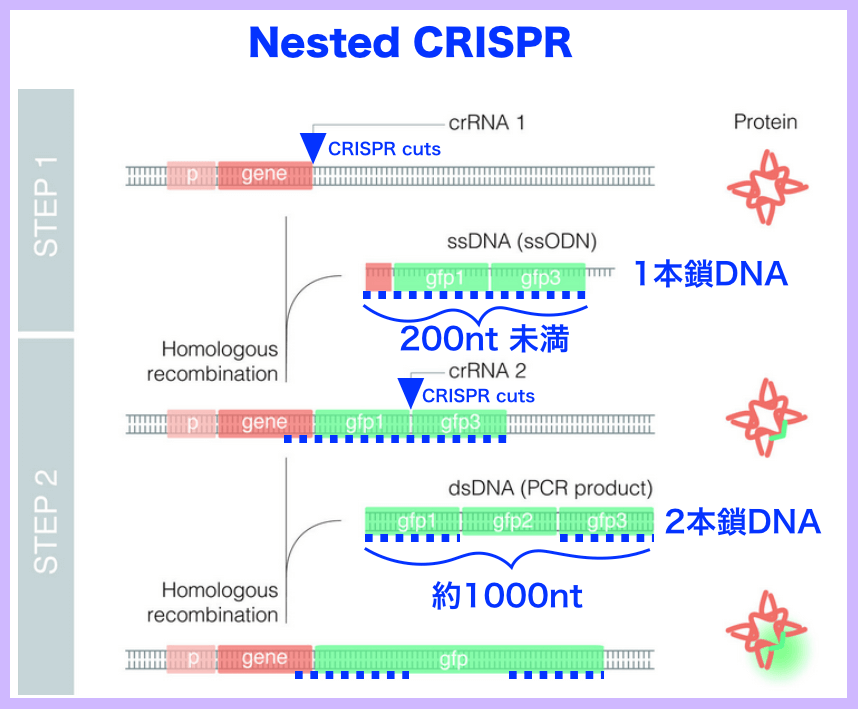
Nested CRISPR、(出典:http://www.idibell.cat) 原図に情報を付け加えました(青色)。(図をクリック/タップすると拡大できます)
STEP 1: 蛍光タンパク質 (GFP)の前側(gfp1)と後側(gfp2)の断片を最初にインフレームでノックインする。効率が良い200 bp未満の1本鎖DNA(ssODN)を使用した。この時STEP 2で利用するCRISPR/Cas9 HDRに必要となるPAMとプロトスペーサを組み込んだ。
STEP 2:残りの蛍光タンパク質断片を含む約1000bpの二本差DNAをテンプレートとしてCRISPR/Cas9 HDRで挿入させた。
※ grRNAではなくcrRNA(もともとCRISPR/Cas9が利用していたガイドRNA)を使った方が効率が良かった。
研究チームは、STEP 1&2に必要な成分を1回のマイクロインジェクションで達成することも可能にし、5種類の遺伝子座のうち3遺伝子座について20%までの編集効率を達成した。
少し前までCRISPRを使って研究していたので、興味がありましたので取り上げてみました。
ノックインされた細胞を作成するのは時間がかかりますが、以上のような方法で効率が上がれば研究コストも下がるので非常にいいですね。

20余年アメリカで遺伝子治療&幹細胞の研究者をやってきました。
特に遺伝病の間違った遺伝子をピンポイントで修復し、元に戻す技術開発です。
理学博士(Ph.D. )
アメリカ生活で学んだアンチエイジングなど健康に関する知識をシェアしたいと思います。
巷の情報もご紹介、時にはブッた斬ったりしてしまうかもしれません。
またポイントなどの節約術や生活に便利なガジェット(スマホやアプリの紹介)も記事にしますので参考になれば嬉しいです。
皆さん、ご一緒に「究極のヘルシーライフ」を楽しみましょう♪
《もっとくわしいプロフィールをみる》



































この記事へのコメントはありません。